逐步回归
逐步回归分析是以AIC信息统计量为准则,通过选择最小的AIC信息统计量,来达到删除或增加变量的目的。
AIC : 赤池信息准则(Akaike Information Criterion) ,k是模型中估计参数的数量。L是模型的似然函数的最大值。
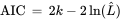
AICc:当样本量很小时,AIC很可能会选择具有太多参数的模型,即AIC会过度拟合。为了解决这种潜在的过度拟合问题,AICc可以对小样本进行校正。其中n表示样本大小,k表示参数的数量。
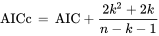
向前逐步回归
首先模型中只有一个单独解释因变量变异最大的自变量,之后尝试将加入另一自变量,看加入后整个模型所能解释的因变量变异是否显著增加(这里需要进行检验,可以用 F-test, t-test 等等)。这一过程反复迭代,直到没有自变量再符合加入模型的条件。
MASS包中的stepAIC()函数给予AIC准则实现了逐步回归模型(向前、向后和双向)。
1 | > library(MASS) |
向后逐步回归
向后逐步回归与向前逐步回归相反,此时,所有变量均放入模型,之后尝试将其中一个自变量从模型中剔除,看整个模型解释因变量的变异是否有显著变化,之后将使解释量减少最少的变量剔除;此过程不断迭代,直到没有自变量符合剔除的条件。
1 | > library(MASS) |
向前向后逐步回归
向前向后逐步回归,这种方法相当于将前两种结合起来。可以想象,如果采用第一种方法,每加入一个自变量,可能会使已存在于模型中的变量单独对因变量的解释度减小,当其的作用很小(不显著)时,则可将其从模型中剔除。而第三种方法就做了这么一件事,不是一味的增加变量,而是增加一个后,对整个模型中的所有变量进行检验,剔除作用不显著的变量。最终尽可能得到一个最优的变量组合。
1 | > library(MASS) |
Forward、Backward、Stepwise的侧重点有所不同,三种方法的选择取决于你的研究目的,如果是进行预测,在预测效果差不多的情况下,一般选择自变量最少的方法。当自变量间不存在多重共线性时,三种方法的计算结果基本一致。当自变量间存在多重共线性时,Forward侧重于引入单独作用较强的变量,Backward侧重于引入联合作用较强的变量,Stepwise介于两者之间。
全子集回归
全子集回归克服了逐步回归的缺点,即所有可能的模型都会被检验,评判准则可以是R平方、修正R平方、BIC或者Mallows Cp统计量。
R平方:

修正R平方 :
我们知道在其他变量不变的情况下,引入新的变量,总能提高模型的R2。修正R2就是相当于给变量的个数加惩罚项。换句话说,如果两个模型,样本数一样,R2一样,那么从修正R2的角度看,使用变量个数少的那个模型更优。其中n是样本数量,p是模型中变量的个数。 当p/n值很小时,如小于0.05,修正R平方将失去修正作用。

BIC:贝叶斯信息准则 ,BIC的惩罚项比AIC的大,考虑了样本数量,样本数量过多时,可有效防止模型精度过高造成的模型复杂度过高。

Mallows Cp:马洛斯的Cp值 与AIC等效。
以下为R中ISLR包的Hitters数据集为例,构建棒球运动员的多元线性模型 。
1 | > library(ISLR) |

交叉验证
交叉验证是在机器学习建立模型和验证模型参数时常用的办法,一般被用于评估一个机器学习模型的表现。更多的情况下,也用交叉验证来进行模型选择(model selection)。
k重交叉验证中,样本被分为k个子样本,轮流将k-1个子样本组合作为训练集,另外1个子样本作为测试集,这样会获得k个预测方程,记录k个测试样本的预测表现结果,然后求其平均值。测试集的目的简单来说就相当于一个游戏的内测,内部评估。进行交叉验证后得到了分数来评估你建模的准确率是高是低。最后的目的是为了哪天来了新的数据,你也可以用你的模型去预测他,相当于游戏公测。
bootstrap包中的crossval() 函数可实现k重交叉验证 :
1 | > install.packages("bootstrap") |